Im folgenden Beispiel setzen wir uns mit Self Organizing Maps (SOM) auseinander.
Self Organinzing Maps sind sehr einfache Künstliche Neuronale Netze. Sie sind in der Lage komplexe, nicht lineare Zusammenhänge, in eine einfache geometrische Beziehungen darzustellen.
Wir werden für unsere Beispiel MiniSom verwenden. Dank dieser Anwednung lassen sich SOM relativ leicht erstellen. Dank gilt an dieser Stelle an Giuseppe Vettigli, da nach meinem Wissen der Entwickler von MiniSom ist. Seine Github Seite ist hier: https://github.com/JustGlowing/minisom
Zu Beginn werden wir die notwendigen Bibliotheken und Daten laden. Wir werden für unser Beispiel den gapminder Datensatz verwenden, den wir etwas anpassen werden. Diese Beispiel wurde in Python programmiert.
#Install gapminder Dataset
pip install gapminder
from gapminder import gapminder
gapminder.head()
export = gapminder.to_excel(r"gapminder.xlsx")
#Install MiniSom
pip install minisom
Wenn man den oben genannten Code laufen lässt hat man den gapminder Datensatz jetzt zur Verfügung und man hat MiniSom installiert. Darüber hinaus hat man den gapminder Datensatz jetzt auch als Excel Datei vorliegen, was man aber nicht unbedingt braucht – jetzt hat man aber auch Code gelernt, der zeigt wie man etwas nach Excel exportieren kann.
Wir laden jetzt die Bibliotheken. Es handel sich hier um die Bibliotheken das fast immer bei Data Science Anwendungen in Python geladen werden. NumPy für scientific computing. pandas zum arbeiten und manipulieren von Daten und Matplotlib zur Datenvisualisierung.
#Import Libraries Import Libraries import numpy as np import matplotlib.pyplot as plt import pandas as pd
Jetzt werden jetzt die gapminder Daten verändern, um Sie in eine Form zu bringen, die es uns erlaubt eine SOM zu berechnen.
#Wrangle with gapminder
dataset = gapminder dataset.head()
dataset = gapminder.groupby("country")["lifeExp","pop","gdpPercap"].mean() dataset.loc[dataset["lifeExp"] < 70, "Nice_Place"] = 0 dataset.loc[dataset["lifeExp"] > 70, "Nice_Place"] = 1 dataset["Nice_Place"] = dataset["Nice_Place"].astype(int) dataset.head(n=20)
X = dataset.iloc[:,0:3]
y = dataset.iloc[:,3]
Durch den Code von oben ist folgendes passiert. Wir haben uns den gapminder Datensatz angesehen und folgendes bemerkt:
country continent year lifeExp pop gdpPercap
0 Afghanistan Asia 1952 28.801 8425333 779.445314
1 Afghanistan Asia 1957 30.332 9240934 820.853030
2 Afghanistan Asia 1962 31.997 10267083 853.100710
3 Afghanistan Asia 1967 34.020 11537966 836.197138
4 Afghanistan Asia 1972 36.088 13079460 739.981106Es ist die jährliche Veränderung der jeweiligen Länder in den Bereichen lifeExp (Lebenserwartung), pop (Population) und gdpPercap (Pro-Kopf Einkommen) aufgelistet. Für unsere SOM benötigen wir aber Durchschnittswerte pro Land. Wir haben dazu die Länder gruppiert und die Durchschnittswerte der einzelnen variablen berechnen lassen. Als nächste wurde eine weitere Spalte in die Tabelle eingefügt um zu erkennen, welche Länder eine Lebenserwartung von über, bzw. unter 70 Jahren haben. Ich habe diese Spalte als „Nice_Place“ bezeichnet, da die Länder mit einer hohen Lebenserwartung vermutlich eine bessere Lebenssituation für deren Einwohner bieten.
„Nice_Pace“ wurde dann noch von float in integer umgewandelt, was auch notwendig zur Erstellung der SOM ist. Als letztes wurden die Daten noch in X (explanatory variable) und y (response variable) eingeteilt. Diese Einteilung ist bei fast allen K.I. / maschinellen Anwendungen notwendig.
Als nächstes werden die Daten skaliert.
#Feature Scaling gapminder from sklearn.preprocessing import MinMaxScaler sc = MinMaxScaler(feature_range = (0,1)) X = sc.fit_transform(X)
Jetzt kann die SOM erstellt bzw. trainiert werden.
#Training the SOM gapminder
from minisom import MiniSom
som = MiniSom(x =14 , y =14 , input_len = 3, sigma = 1.0,
learning_rate = 0.5 )
som.random_weights_init(X)
som.train_random(data = X , num_iteration = 1000)
Als nächstes folgt der Code zur visuellen Darstellung der Ergebnisse.
#Visualize the Results gapminder
from pylab import bone, pcolor, colorbar, plot, show
bone()
pcolor(som.distance_map().T)
colorbar()
markers = ["o","s"]
colors = ["r","g"]
for i, x in enumerate(X):
w = som.winner(x)
plot(w[0] + 0.5,
w[1] + 0.5,
markers[y[i]],
markeredgecolor = colors[y[i]],
markerfacecolor = "None",
markersize = 10,
markeredgewidth = 2)
show()
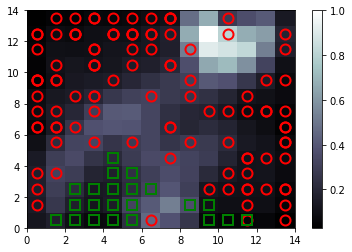
Man erkennt, dass Länder mit einer Lebenserwartung von über 70 Jahren (grüne Vierecke) alle vom neuronalen netz, nahe beieinander angeordnet wurden. Sie sind also ähnlich. Das Land bei X = 6 und Y = 0 ist übrigens Kuwait. Es ist nahe an den Ländern mit einer hohen Lebenserwartung. Die Erklärung ist folgende: Kuwait hat ein relativ hohes Pro Kopf Einkommen (65332,91) – was sicherlich westlicher Standard ist, allerdings liegt die Lebenserwartung bei 68,9 Jahren. In diesem Land scheint man reich zu sein aber früh zu sterben. Man muss aber bedenken, dass es sich hier um Durchschnittswerte handelt. Man könnte hier z.B. die Hypothese erstellen, dass es sich um ein Land handelt mit einer relativ kleinen Elite von super reichen und vielen armen normal Bürger.

Schreibe einen Kommentar