In diesem Artikel möchten wir die Hierarchische Clusteranalyse vorstellen.
Ist gibt zwei grundsätzliche Verfahren für die Hierarchische Clusteranalyse. Man nennt sie Top-Down und Bottom-Up Verfahren.
Die Top-Down Verfahren werden als divisive Cluster-verfahren bezeichnet. Im englischen Sprachgebrauch wird hierfür gerne die Abkürzung DIANA verwendet. Sie steht für Divise Analysis.
Wir steigen gleich ein, indem wir die notwendigen Bibliotheken laden:
library(tidyverse) # data manipulation library(cluster) # clustering algorithms library(factoextra) # clustering visualization library(gapminder)
Verwendet wird in unserem Beispiel der gapminder Datensatz. Wir haben diesen bereits mit library(gapminder) geladen.
Betrachten wir den Datensatz mit der funktion head().
head()
A tibble: 6 x 6
country continent year lifeExp pop gdpPercap
1 Afghanistan Asia 1952 28.8 8425333 779.
2 Afghanistan Asia 1957 30.3 9240934 821.
3 Afghanistan Asia 1962 32.0 10267083 853.
4 Afghanistan Asia 1967 34.0 11537966 836.
5 Afghanistan Asia 1972 36.1 13079460 740.
6 Afghanistan Asia 1977 38.4 14880372 786.
Der gapminder Datensatz zeigt die Entwicklungen der einzelnen Länder pro Jahr. Da wir Länder untereinander vergleichen möchten, werden wir mit Durchschnittswerten arbeiten. Diese erzeugen wir mit folgenden Code:
df <- gapminder %>%
group_by(country) %>%
summarise(lifeExp_mean = mean(lifeExp),
pop_mean = mean(pop),
gdpPercap_mean = mean(gdpPercap)) %>%
filter(lifeExp_mean < 50) %>%
na.omit()
head(df)
A tibble: 6 x 4
country lifeExp_mean pop_mean gdpPercap_mean
1 Afghanistan 37.5 15823715. 803.
2 Albania 68.4 2580249. 3255.
3 Algeria 59.0 19875406. 4426.
4 Angola 37.9 7309390. 3607.
5 Argentina 69.1 28602240. 8956.
6 Australia 74.7 14649312. 19981.
Mit den nächsten Schritten werden wir die Daten anpassen, damit man eine Hierarchische Clusteranalyse durchführen kann. Zu Beginn werden wir die erste Spalte umwandeln, damit diese als Reihen namen erkannt wird.
df <- column_to_rownames(df,"country")
head(df)
lifeExp_mean pop_mean gdpPercap_mean
Afghanistan 37.47883 15823715 802.6746
Albania 68.43292 2580249 3255.3666
Algeria 59.03017 19875406 4426.0260
Angola 37.88350 7309390 3607.1005
Argentina 69.06042 28602240 8955.5538
Australia 74.66292 14649313 19980.5956
Danach folgt die Skalierung/Standardisierung der Daten mit scale().
df <- scale(df) d <- dist(df, method = "euclidean") head(d)
Nachdem die Skalierung / Standardisierung abgeschlossen ist, werden die Daten auf Unterschiede untersucht, bzw. es wird eine Distanzmatrix erstellt. Wir verwenden in unserem Beispiel den häufig verwendeten euklidischen Abstand. Wir haben die dist() funktion bereits oben ausgeführt.
Die Distanzmatrix lässt sich auch bildlich darstellen. Wir verwenden dazu folgende Code:
fviz_dist(d, gradient = list(low = "white", mid = "blue", high = "red"))
Dadurch erhält man folgende Distanzmatrix als Bild:
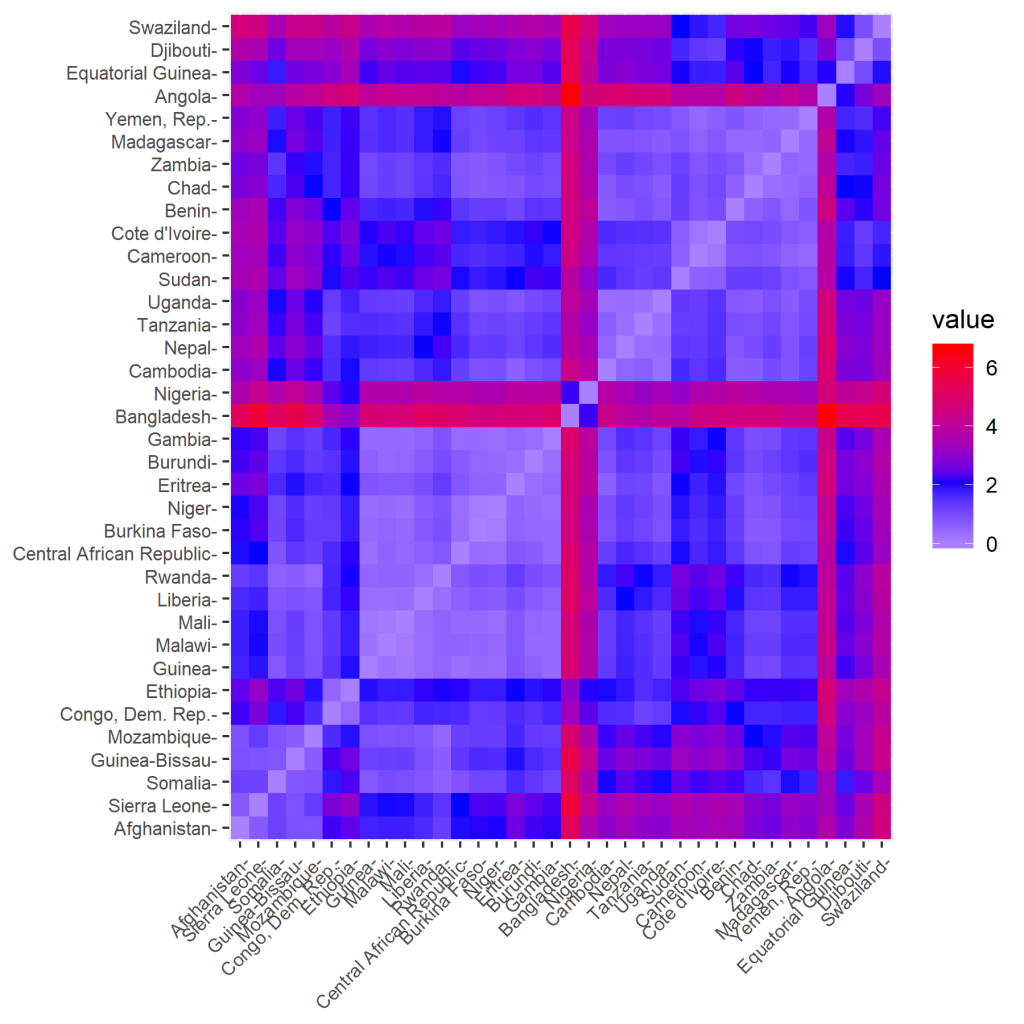
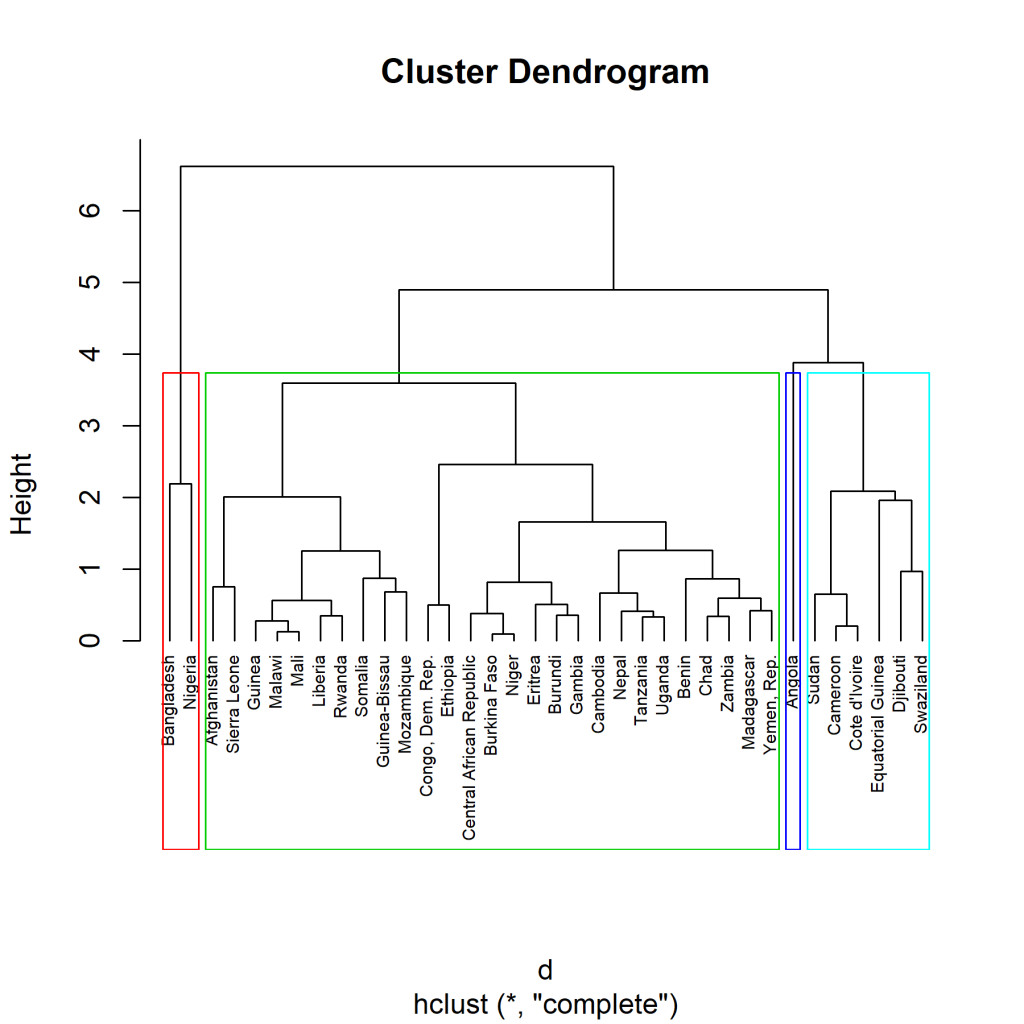
Jetzt führen wir die Berechnung der Cluster durch. Dazu verwenden wir die „complete“ Methode.
hc1 <- hclust(d, method = "complete" ) plot(hc1, cex = 0.6, hang = -1) rect.hclust(hc1, k = 4, border = 2:6)
Mit der Funktion rect.hclust() haben wir die Cluster noch farblich in Quadrate eingeteilt.
Eine weitere Möglichkeit ist der Verwendung des factoextra Packetes. Wie führen den folgenden Code aus um das Paket zu verwenden:
hc.cut <- hcut(d, k = 4, hc_method = "complete") fviz_dend(hc.cut, show_labels = TRUE, rect = TRUE) fviz_cluster(data=d,hc.cut, frame.type = "convex")
Erstell wird dadurch ein weiteres Dendrogramm und ein weitere Darstellung der Cluster.
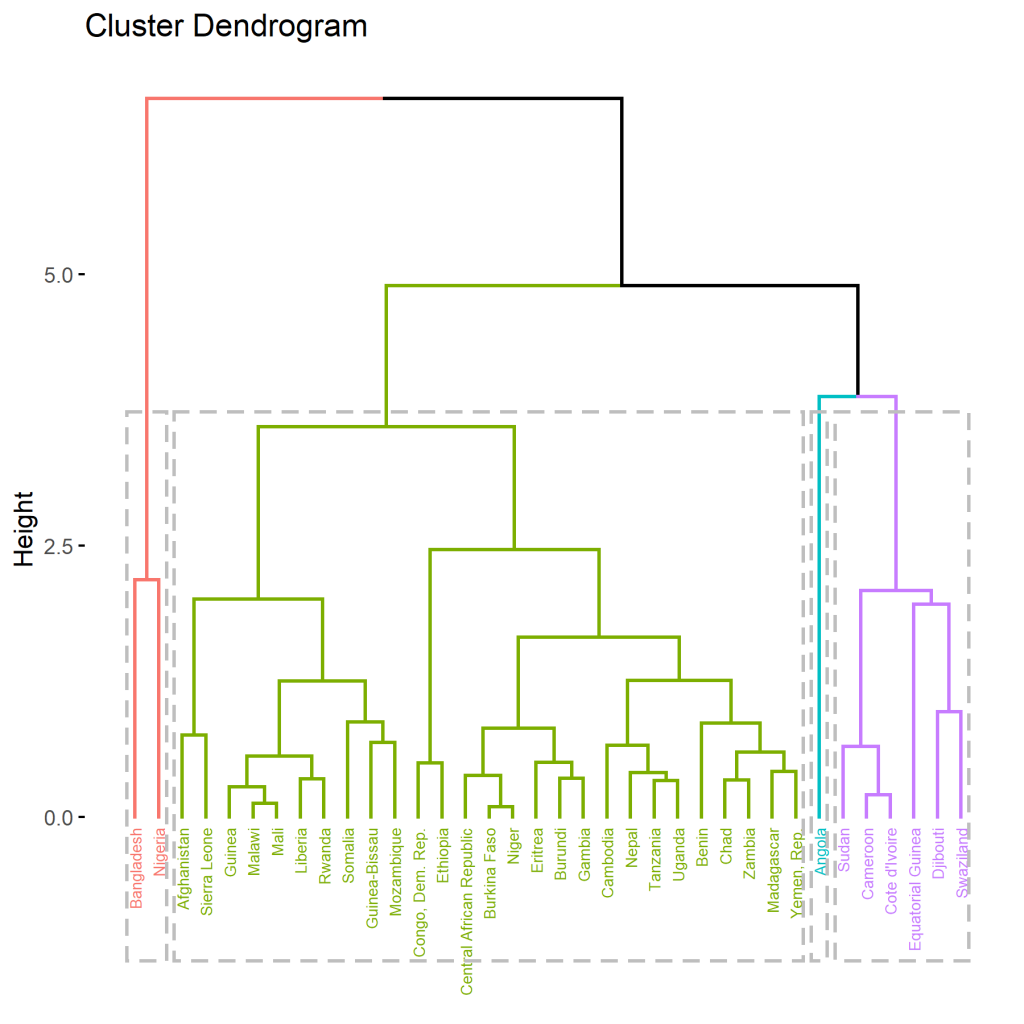
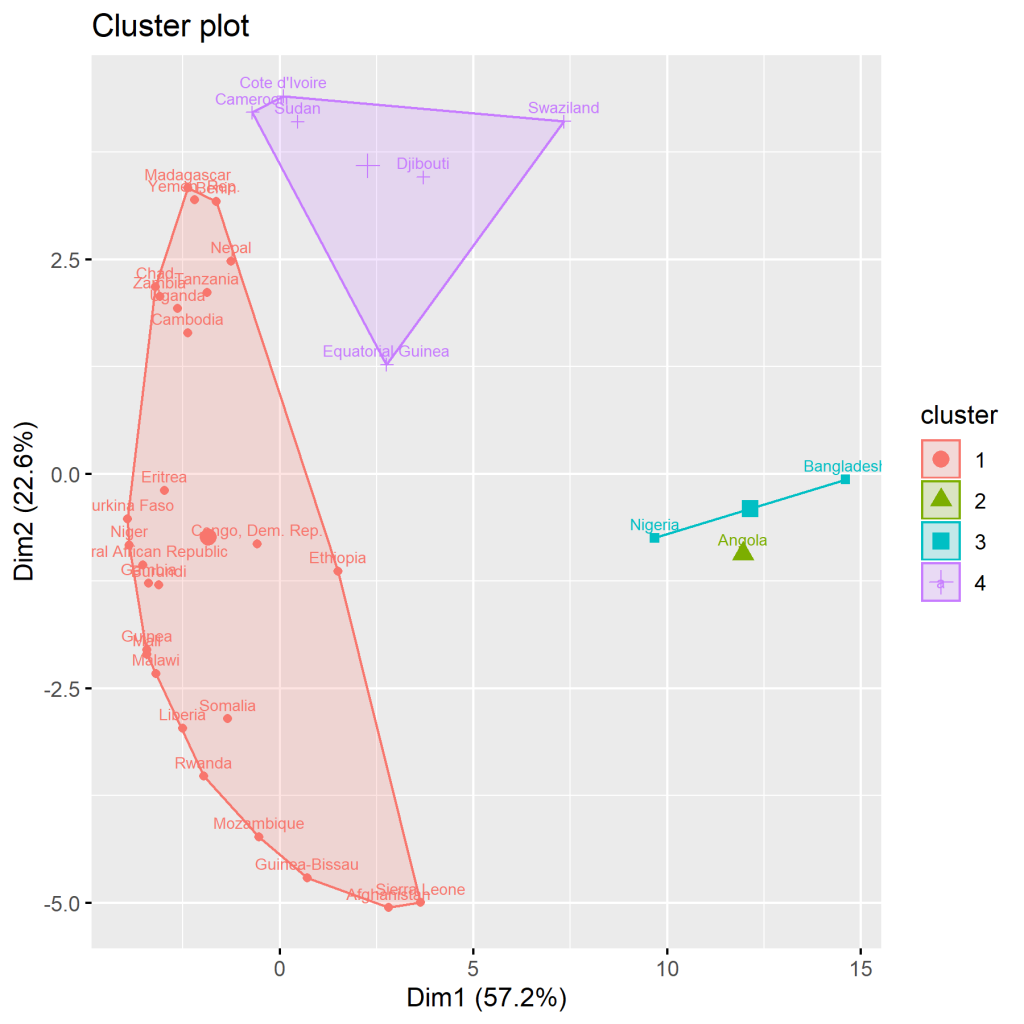

Schreibe einen Kommentar